Concurso:
IF Sertão - PE
Disciplina:
Biologia
A figura a seguir apresenta um esquema resumido das etapas que levam à síntese de proteínas a partir do gene em eucariotos, indicando diferentes etapas do processamento do RNA, incluindo a adição do cap 5', a poliadenilação e o splicing. Com base na figura e sobre a biologia molecular no processo de splicing do RNA, analise as assertivas abaixo e assinale V, se verdadeiras, ou F, se falsas.
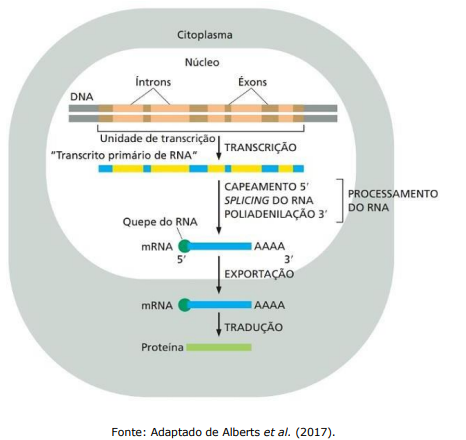
( ) A remoção dos íntrons do pré-mRNA ocorre por meio de duas reações de transesterificação catalisadas por moléculas de RNA (snRNA) e não por proteínas, tornando o splicing uma das raras reações enzimáticas em que o próprio RNA atua como catalisador.
( ) Apesar da variabilidade nas sequências consenso que definem os limites entre éxons e íntrons, a célula é capaz de identificar corretamente os locais de splicing com base exclusivamente nessas sequências, sem a necessidade de proteínas auxiliares ou outras estratégias regulatórias.
( ) A função evolutiva do splicing alternativo ainda é considerada limitada, uma vez que combinações distintas de éxons a partir de um mesmo gene geralmente não resultam em variações significativas na função das proteínas produzidas.
( ) A precisão do splicing é favorecida por dois mecanismos principais: (I) a coordenação espacial e temporal (acoplamento) entre transcrição e splicing e (II) a definição de éxons baseada em sua uniformidade de tamanho e na marcação por proteínas SR, que recrutam os snRNAs U1 e U2.
( ) A marcação dos limites entre éxons e íntrons, a montagem do spliceossomo e a remoção dos íntrons ocorrem de forma cotranscricional e linear, seguindo a ordem em que os íntrons surgem durante a síntese do pré-mRNA. Desse modo, cada íntron é removido assim que transcrito de forma linear, permitindo uma maior precisão regulatória no processamento do transcrito.
A ordem correta de preenchimento dos parênteses, de cima para baixo, é:

( ) A remoção dos íntrons do pré-mRNA ocorre por meio de duas reações de transesterificação catalisadas por moléculas de RNA (snRNA) e não por proteínas, tornando o splicing uma das raras reações enzimáticas em que o próprio RNA atua como catalisador.
( ) Apesar da variabilidade nas sequências consenso que definem os limites entre éxons e íntrons, a célula é capaz de identificar corretamente os locais de splicing com base exclusivamente nessas sequências, sem a necessidade de proteínas auxiliares ou outras estratégias regulatórias.
( ) A função evolutiva do splicing alternativo ainda é considerada limitada, uma vez que combinações distintas de éxons a partir de um mesmo gene geralmente não resultam em variações significativas na função das proteínas produzidas.
( ) A precisão do splicing é favorecida por dois mecanismos principais: (I) a coordenação espacial e temporal (acoplamento) entre transcrição e splicing e (II) a definição de éxons baseada em sua uniformidade de tamanho e na marcação por proteínas SR, que recrutam os snRNAs U1 e U2.
( ) A marcação dos limites entre éxons e íntrons, a montagem do spliceossomo e a remoção dos íntrons ocorrem de forma cotranscricional e linear, seguindo a ordem em que os íntrons surgem durante a síntese do pré-mRNA. Desse modo, cada íntron é removido assim que transcrito de forma linear, permitindo uma maior precisão regulatória no processamento do transcrito.
A ordem correta de preenchimento dos parênteses, de cima para baixo, é: